8 Example: Microarray data visualization
ต่อไปนี้จะเป็นตัวอย่างในการใช้ R ในการวิเคราะห์ข้อมูล Molecular data เบื้องต้น โดยใช้ข้อมูลจาก GEO dataset GSE63514 Den Boon et al. (2015) ซึ่งเป็น Gene expression microarray

8.1 Import files
ไฟล์แรกที่อ่านเข้ามาคือ GSE63514_norm.csv ซึ่งเป็นไฟล์ Microarray expression ของชิ้นเนื้อปากมดลูก ประกอบด้วย ตัวอย่าง Normal , CIN1 , CIN2 , CIN3 , Cancer ในที่นี้เราจะทำการวิเคราะห์ 20,000 ยีนแรก
library(tidyverse)
GSE63514 <- read_csv("Resource/GSE63514_norm.csv")
GSE63514_20000 <- read_csv("Resource/GSE63514_norm.csv") |> head(2000)
names(GSE63514_20000)## [1] "probe" "GSM1551311_Normal-01_U133_Plus2.CEL.gz"
## [3] "GSM1551312_Normal-02_U133_Plus2.CEL.gz" "GSM1551313_Normal-03_U133_Plus2.CEL.gz"
## [5] "GSM1551314_Normal-04_U133_Plus2.CEL.gz" "GSM1551315_Normal-05_U133_Plus2.CEL.gz"
## [7] "GSM1551316_Normal-06_U133_Plus2.CEL.gz" "GSM1551317_Normal-07_U133_Plus2.CEL.gz"
## [9] "GSM1551318_Normal-08_U133_Plus2.CEL.gz" "GSM1551319_Normal-09_U133_Plus2.CEL.gz"
## [11] "GSM1551320_Normal-10_U133_Plus2.CEL.gz" "GSM1551321_Normal-11_U133_Plus2.CEL.gz"
## [13] "GSM1551322_Normal-12_U133_Plus2.CEL.gz" "GSM1551323_Normal-13_U133_Plus2.CEL.gz"
## [15] "GSM1551324_Normal-14_U133_Plus2.CEL.gz" "GSM1551325_Normal-15_U133_Plus2.CEL.gz"
## [17] "GSM1551326_Normal-16_U133_Plus2.CEL.gz" "GSM1551327_Normal-17_U133_Plus2.CEL.gz"
## [19] "GSM1551328_Normal-18_U133_Plus2.CEL.gz" "GSM1551329_Normal-19_U133_Plus2.CEL.gz"
## [21] "GSM1551330_Normal-20_U133_Plus2.CEL.gz" "GSM1551331_Normal-21_U133_Plus2.CEL.gz"
## [23] "GSM1551332_Normal-22_U133_Plus2.CEL.gz" "GSM1551333_Normal-23_U133_Plus2.CEL.gz"
## [25] "GSM1551334_Normal-24_U133_Plus2.CEL.gz" "GSM1551335_CIN1-01_U133_Plus2.CEL.gz"
## [27] "GSM1551336_CIN1-02_U133_Plus2.CEL.gz" "GSM1551337_CIN1-03_U133_Plus2.CEL.gz"
## [29] "GSM1551338_CIN1-04_U133_Plus2.CEL.gz" "GSM1551339_CIN1-05_U133_Plus2.CEL.gz"
## [31] "GSM1551340_CIN1-06_U133_Plus2.CEL.gz" "GSM1551341_CIN1-07_U133_Plus2.CEL.gz"
## [33] "GSM1551342_CIN1-08_U133_Plus2.CEL.gz" "GSM1551343_CIN1-09_U133_Plus2.CEL.gz"
## [35] "GSM1551344_CIN1-10_U133_Plus2.CEL.gz" "GSM1551345_CIN1-11_U133_Plus2.CEL.gz"
## [37] "GSM1551346_CIN1-12_U133_Plus2.CEL.gz" "GSM1551347_CIN1-13_U133_Plus2.CEL.gz"
## [39] "GSM1551348_CIN1-14_U133_Plus2.CEL.gz" "GSM1551349_CIN2-01_U133_Plus2.CEL.gz"
## [41] "GSM1551350_CIN2-02_U133_Plus2.CEL.gz" "GSM1551351_CIN2-03_U133_Plus2.CEL.gz"
## [43] "GSM1551352_CIN2-04_U133_Plus2.CEL.gz" "GSM1551353_CIN2-05_U133_Plus2.CEL.gz"
## [45] "GSM1551354_CIN2-06_U133_Plus2.CEL.gz" "GSM1551355_CIN2-07_U133_Plus2.CEL.gz"
## [47] "GSM1551356_CIN2-08_U133_Plus2.CEL.gz" "GSM1551357_CIN2-09_U133_Plus2.CEL.gz"
## [49] "GSM1551358_CIN2-10_U133_Plus2.CEL.gz" "GSM1551359_CIN2-11_U133_Plus2.CEL.gz"
## [51] "GSM1551360_CIN2-12_U133_Plus2.CEL.gz" "GSM1551361_CIN2-13_U133_Plus2.CEL.gz"
## [53] "GSM1551362_CIN2-14_U133_Plus2.CEL.gz" "GSM1551363_CIN2-15_U133_Plus2.CEL.gz"
## [55] "GSM1551364_CIN2-16_U133_Plus2.CEL.gz" "GSM1551365_CIN2-17_U133_Plus2.CEL.gz"
## [57] "GSM1551366_CIN2-18_U133_Plus2.CEL.gz" "GSM1551367_CIN2-19_U133_Plus2.CEL.gz"
## [59] "GSM1551368_CIN2-20_U133_Plus2.CEL.gz" "GSM1551369_CIN2-21_U133_Plus2.CEL.gz"
## [61] "GSM1551370_CIN2-22_U133_Plus2.CEL.gz" "GSM1551371_CIN3-01_U133_Plus2.CEL.gz"
## [63] "GSM1551372_CIN3-02_U133_Plus2.CEL.gz" "GSM1551373_CIN3-03_U133_Plus2.CEL.gz"
## [65] "GSM1551374_CIN3-04_U133_Plus2.CEL.gz" "GSM1551375_CIN3-05_U133_Plus2.CEL.gz"
## [67] "GSM1551376_CIN3-06_U133_Plus2.CEL.gz" "GSM1551377_CIN3-07_U133_Plus2.CEL.gz"
## [69] "GSM1551378_CIN3-08_U133_Plus2.CEL.gz" "GSM1551379_CIN3-09_U133_Plus2.CEL.gz"
## [71] "GSM1551380_CIN3-10_U133_Plus2.CEL.gz" "GSM1551381_CIN3-11_U133_Plus2.CEL.gz"
## [73] "GSM1551382_CIN3-12_U133_Plus2.CEL.gz" "GSM1551383_CIN3-13_U133_Plus2.CEL.gz"
## [75] "GSM1551384_CIN3-14_U133_Plus2.CEL.gz" "GSM1551385_CIN3-15_U133_Plus2.CEL.gz"
## [77] "GSM1551386_CIN3-16_U133_Plus2.CEL.gz" "GSM1551387_CIN3-17_U133_Plus2.CEL.gz"
## [79] "GSM1551388_CIN3-18_U133_Plus2.CEL.gz" "GSM1551389_CIN3-19_U133_Plus2.CEL.gz"
## [81] "GSM1551390_CIN3-20_U133_Plus2.CEL.gz" "GSM1551391_CIN3-21_U133_Plus2.CEL.gz"
## [83] "GSM1551392_CIN3-22_U133_Plus2.CEL.gz" "GSM1551393_CIN3-23_U133_Plus2.CEL.gz"
## [85] "GSM1551394_CIN3-24_U133_Plus2.CEL.gz" "GSM1551395_CIN3-25_U133_Plus2.CEL.gz"
## [87] "GSM1551396_CIN3-26_U133_Plus2.CEL.gz" "GSM1551397_CIN3-27_U133_Plus2.CEL.gz"
## [89] "GSM1551398_CIN3-28_U133_Plus2.CEL.gz" "GSM1551399_CIN3-29_U133_Plus2.CEL.gz"
## [91] "GSM1551400_CIN3-30_U133_Plus2.CEL.gz" "GSM1551401_CIN3-31_U133_Plus2.CEL.gz"
## [93] "GSM1551402_CIN3-32_U133_Plus2.CEL.gz" "GSM1551403_CIN3-33_U133_Plus2.CEL.gz"
## [95] "GSM1551404_CIN3-34_U133_Plus2.CEL.gz" "GSM1551405_CIN3-35_U133_Plus2.CEL.gz"
## [97] "GSM1551406_CIN3-36_U133_Plus2.CEL.gz" "GSM1551407_CIN3-37_U133_Plus2.CEL.gz"
## [99] "GSM1551408_CIN3-38_U133_Plus2.CEL.gz" "GSM1551409_CIN3-39_U133_Plus2.CEL.gz"
## [101] "GSM1551410_CIN3-40_U133_Plus2.CEL.gz" "GSM1551411_Cancer-01_U133_Plus2.CEL.gz"
## [103] "GSM1551412_Cancer-02_U133_Plus2.CEL.gz" "GSM1551413_Cancer-03_U133_Plus2.CEL.gz"
## [105] "GSM1551414_Cancer-04_U133_Plus2.CEL.gz" "GSM1551415_Cancer-05_U133_Plus2.CEL.gz"
## [107] "GSM1551416_Cancer-06_U133_Plus2.CEL.gz" "GSM1551417_Cancer-07_U133_Plus2.CEL.gz"
## [109] "GSM1551418_Cancer-08_U133_Plus2.CEL.gz" "GSM1551419_Cancer-09_U133_Plus2.CEL.gz"
## [111] "GSM1551420_Cancer-10_U133_Plus2.CEL.gz" "GSM1551421_Cancer-11_U133_Plus2.CEL.gz"
## [113] "GSM1551422_Cancer-12_U133_Plus2.CEL.gz" "GSM1551423_Cancer-13_U133_Plus2.CEL.gz"
## [115] "GSM1551424_Cancer-14_U133_Plus2.CEL.gz" "GSM1551425_Cancer-15_U133_Plus2.CEL.gz"
## [117] "GSM1551426_Cancer-16_U133_Plus2.CEL.gz" "GSM1551427_Cancer-17_U133_Plus2.CEL.gz"
## [119] "GSM1551428_Cancer-18_U133_Plus2.CEL.gz" "GSM1551429_Cancer-19_U133_Plus2.CEL.gz"
## [121] "GSM1551430_Cancer-20_U133_Plus2.CEL.gz" "GSM1551431_Cancer-21_U133_Plus2.CEL.gz"
## [123] "GSM1551432_Cancer-22_U133_Plus2.CEL.gz" "GSM1551433_Cancer-23_U133_Plus2.CEL.gz"
## [125] "GSM1551434_Cancer-24_U133_Plus2.CEL.gz" "GSM1551435_Cancer-25_U133_Plus2.CEL.gz"
## [127] "GSM1551436_Cancer-26_U133_Plus2.CEL.gz" "GSM1551437_Cancer-27_U133_Plus2.CEL.gz"
## [129] "GSM1551438_Cancer-28_U133_Plus2.CEL.gz"ส่วนที่สองคือ Metadata (ข้อมูลระบุตัวตน) ของข้อมูลนี้
head(GSE63514_meta, 10)ส่วนที่สามคือไฟล์ Annotation ของ Probe
hgu133plus2_genenames <- read_csv("Resource/hgu133plus2_genenames.csv") |>
select(-1) # remove row number
head(hgu133plus2_genenames, 10)8.2 Cleaning data
สมมติว่าในตัวอย่างนี้ จะทำการวิเคราะห์แค่ระหว่าง Normal, CIN1, และ Cancer เท่านั้น จึงจำเป็นที่จะต้องกรองข้อมูลที่ไม่ต้องการออกไปเสียก่อน
exprs_nc <- GSE63514_20000 |> select(probe, contains(c("Normal", "CIN1", "Cancer")))
meta_nc <- GSE63514_meta |>
filter(grepl("Normal|CIN1|Cancer", title)) |>
select(title, `characteristics_ch1.1`, `dissection:ch1`)
names(exprs_nc) <- c("prob", meta_nc$title) # เปลี่ยนชื่อให้อ่านง่าย
head(exprs_nc, 10)
head(meta_nc, 10)เมื่อดูในตัวแปร exps_nc จะพบว่า probe นั้นเป็นชื่อเฉพาะของตัวเครื่อง ไม่ใช้ชื่อสากล ในที่นี้จะทำการเปลี่ยน Probe ให้เป็นชื่อยีนนั้นๆ แต่ว่าชื่อ List รายชื่อนั้นเป็นชื่อทั้งหมดของ Probe สังเกตได้จากจำนวนแถวที่ไม่เท่ากัน
nrow(exprs_nc)## [1] 2000
nrow(hgu133plus2_genenames)## [1] 54675ในที่นี้ การใช้คำสั่ง *_join() จะทำให้สามารถรวมแค่แถวที่ต้องการได้
8.3 Visualization
ต่อไป จะทำการแสดงผล Gene expression 10 ตัวที่มีการแสดงออกมากที่สุดในทั้งการทดลองนี้
ขั้นแรก เราจะทำการรวม Intensity ทั้งหมดใน 1 ยีน ผ่านฟังก์ชัน rowSums() และเรียง total_intensity จากมากไปน้อย หลังจากนั้นเลือก Top 10 intensity ออกมาโดยใช้ function head()
gene_symbol_mat <- gene_nc |>
select(-prob, -ENTREZID, -GENENAME) |>
mutate(total_intensity =
rowSums(select(gene_nc, -prob, -ENTREZID, -GENENAME, -SYMBOL)),
.before = "Normal-01") |>
arrange(desc(total_intensity))
top10_intensity <- head(gene_symbol_mat,10)
top10_intensityจะเห็นว่าข้อมูลของเรา อยู่ในลักษณะ wide form ในการสร้าง boxplot นั้น ข้อมูลจำเป็นต้องอยู่ในลักษณะ long form เราจะใช้ function pivot_longer()
top10_long <- top10_intensity |>
select(-total_intensity) |>
pivot_longer(-SYMBOL, names_to = "Case",
values_to = "Intensity") |>
separate(Case, into = c("Group", "Number"), sep = "-", remove = FALSE)
ggplot(top10_long, aes(x = SYMBOL, y = Intensity, fill = Group)) +
geom_boxplot() +
theme_bw() +
theme(axis.text.x =
element_text(angle = 45, vjust = 1, hjust=1)) # หมุนแกน x เพื่อความสวยงาม
ท่านอาจจะอยากเพิ่มเส้นค่าเฉลี่ยเพื่อดูว่า Global mean intensity เป็นเท่าไร
global_mean_intensity <- mean(top10_long$Intensity)
global_mean_intensity## [1] 12.99896
ggplot(top10_long, aes(x = SYMBOL, y = Intensity, fill = Group)) +
geom_boxplot() +
geom_hline(yintercept = global_mean_intensity, linetype = "dashed", color = "darkviolet", linewidth = 1) +
theme_bw() +
theme(axis.text.x =
element_text(angle = 45, vjust = 1, hjust=1))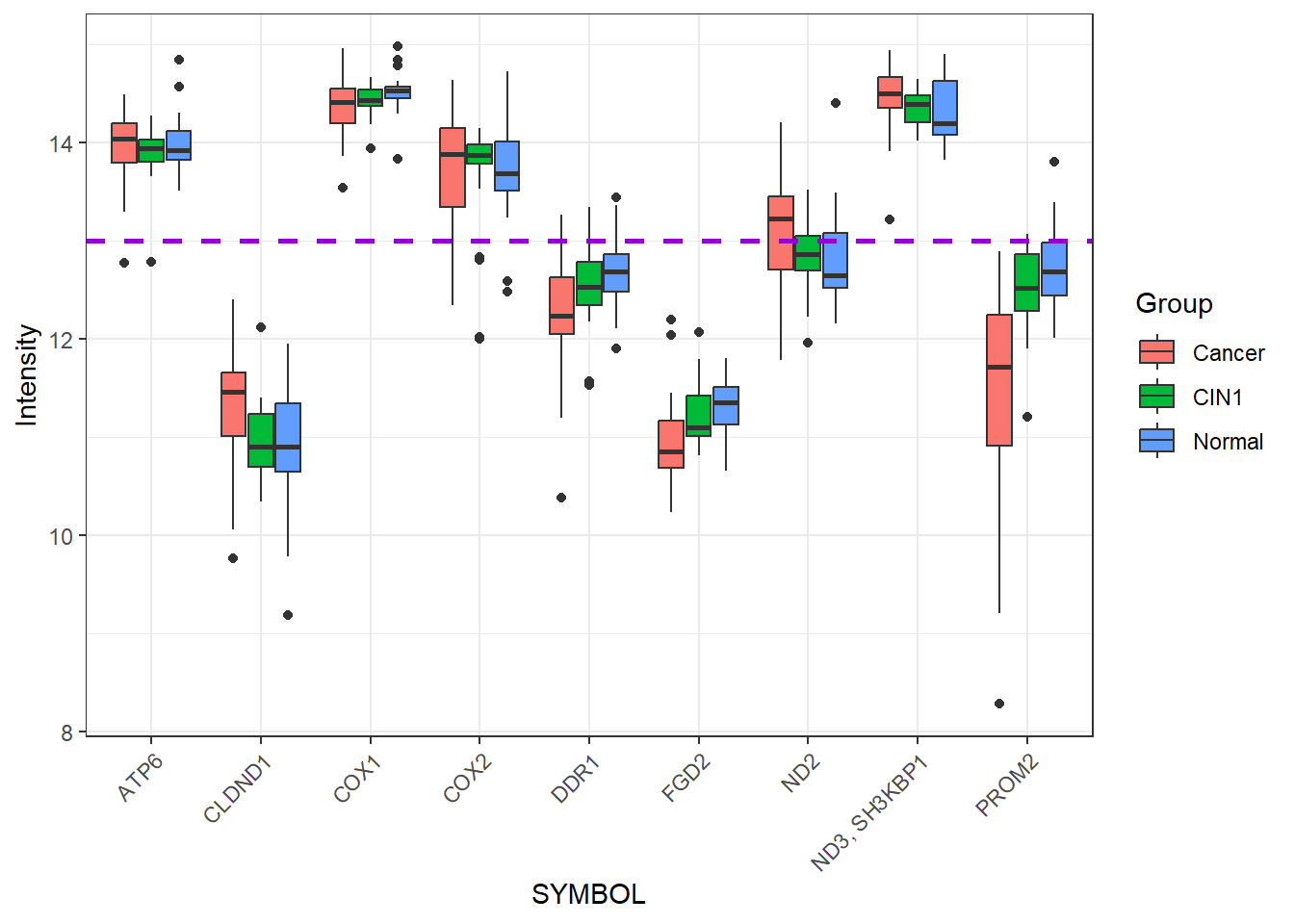
ตัวกลุ่ม RPL ดูน่าสนใน เนื่องจาก Intensity ใน cancer ต่ำกว่า global mean แต่จะมีนัยสำคัญหรือไม่ต้องใช้การวิเคราะห์ทางสถิติเพิ่มเติมทีหลัง
ต่อไป เราอยากที่จะแบ่งว่า tissue ที่เป็น whole section กับ laser captured มีการแสดงออกที่แตกต่างกันอย่างไร เราจะใช้ facet_wrap() เข้ามาช่วย
top10_long_dissec <- top10_long |>
left_join(select(meta_nc, title, `dissection:ch1`),
by = c("Case" = "title"))
top10_long_dissecพล็อตอัตราส่วนจำนวนของ laser capture vs whole section
ggplot(top10_long_dissec, aes(x = `dissection:ch1`, fill = Group)) +
geom_bar(col = "black", width = 0.5, position = "dodge") +
theme_bw()
เห็นว่าผลที่ได้ประหลาด เนื่องจาก laser captured และ laser-captured เป็นตัวแปรซ้ำ ต้องแก้ไขเสียก่อน
top10_long_dissec <- top10_long_dissec |>
mutate(`dissection:ch1` = gsub("-", " ", `dissection:ch1`))
ggplot(top10_long_dissec, aes(x = `dissection:ch1`, fill = Group)) +
geom_bar(col = "black", width = 0.5, position = "dodge") +
theme_bw()
เมื่อข้อมูลที่ได้ถูกต้อง จะเห็นว่า มีเฉพาะกลุ่ม cancer เท่านั้น ที่มีการตัดแบบ whole section
หลังจากนั้นเราจะทำการพล็อต Intensity ในแต่ละยีน
ggplot(top10_long_dissec, aes(x = SYMBOL, y = Intensity, fill = Group)) +
geom_boxplot() +
facet_wrap(~`dissection:ch1`) +
theme_bw() +
theme(axis.text.x =
element_text(angle = 45, vjust = 1, hjust=1))