15 Principal component analysis
15.1 Principle
Principal component analysis (PCA) คือ วิธีการลดจำนวนมิติของข้อมูล (Dimensional reduction) เพื่อให้ง่ายต่อการวิเคราะห์และสร้างภาพ โดยที่ PCA จะพยายามเก็บข้อมูลที่สำคัญไว้
ยกตัวอย่างเป็นภาพ สมมติท่านต้องการที่จะวิเคราะห์แบ่งลักษณะของรูปจากสีของรูปภาพ แต่เฉดสีนั้นมีมากมาย

ถ้าเป็นข้อมูลอาจจะมีรูปแบบลักษณะนี้
Picture_A <dbl> | Picture_B <dbl> | Picture_C <dbl> | Picture_D <dbl> | Picture_E <dbl> | Picture_F <dbl> | Picture_G <dbl> | Picture_H <dbl> | Picture_I <dbl> | ||
|---|---|---|---|---|---|---|---|---|---|---|
| PaleRed | -2.0686670 | 7.253815 | 9.49281009 | 6.338540 | 8.273959 | 4.47989502 | 13.7231198 | 18.5045004 | -0.7646529 | |
| LightRed | 6.7184877 | 6.426464 | -1.28027292 | 7.995346 | 11.823893 | 6.81820714 | 2.7494272 | 12.5447722 | -1.8922639 | |
| MediumRed | 2.6731021 | 5.704929 | 9.74354898 | 10.290517 | 5.146352 | 0.11221368 | 4.8946828 | 12.6809739 | -3.9450925 | |
| DarkRed | 2.6482741 | 6.895126 | 2.03324093 | 8.305544 | 9.833314 | 4.40356867 | 5.9097090 | 15.8288828 | -4.1388629 | |
| VerydarkRed | 3.5492478 | 6.878133 | 1.12719809 | 6.022205 | 11.558222 | 2.47560502 | 2.5486581 | 6.1186805 | -1.2958939 | |
| PaleBlue | 6.8416646 | 6.821581 | -0.09191291 | 10.446423 | 10.903460 | -0.14428832 | 5.5505861 | 4.9763384 | 3.4719433 | |
| LightBlue | -2.1818119 | 6.688640 | 11.59967734 | 9.980512 | 10.317029 | -0.02933531 | 5.1068936 | 6.3246709 | 5.4393926 | |
| MediumBlue | 8.0705531 | 6.553918 | -2.33741451 | 8.645191 | 8.077882 | -0.20307235 | 3.3538585 | 12.9577395 | 3.8303534 | |
| DarkBlue | 4.5114373 | 5.938088 | 4.98348192 | 7.716195 | 7.450887 | 1.93818696 | 4.1958791 | 5.4619642 | -0.4546292 | |
| VerydarkBlue | 10.5850097 | 5.694037 | 7.93590496 | 5.116282 | 6.927614 | 0.07648883 | 3.8977982 | 2.6222831 | 1.2389997 |
ซึ่งการที่จะนำข้อมูลสีทุกจุดมาวิเคราะห์นั้น อาจจะต้องใช้เครื่องคอมพิวเตอร์ที่มีสมรรถนะสูงมาก และข้อมูลส่วนใหญ่ก็ไม่ได้ต่างกันมากนัก ท่านจึงตัดสินใจรวมสีที่มีลักษณะใกล้เคียงกันเป็นกลุ่มๆ
.png)
จากกระบวนการนี้ รวมสีที่ใกล้กันเป็นกลุ่มเดียว ส่งผลให้เกิดการลดจำนวนมิติของข้อมูลลง โดยที่ยังเหลือข้อมูลที่มีความสำคัญอยู่
Picture_A <dbl> | Picture_B <dbl> | Picture_C <dbl> | Picture_D <dbl> | Picture_E <dbl> | Picture_F <dbl> | Picture_G <dbl> | Picture_H <dbl> | Picture_I <dbl> | ||
|---|---|---|---|---|---|---|---|---|---|---|
| Red | -2.068667 | 10.070553 | 11.294763 | 4.869978 | 7.253815 | 11.769588 | 6.876891 | 8.885813 | 8.274931 | |
| Blue | 6.718488 | 6.511437 | 10.825540 | 6.355544 | 6.426464 | 8.690441 | 7.597115 | 8.828518 | 7.595399 | |
| Green | 2.673102 | 12.585010 | 9.944314 | 6.874804 | 5.704929 | 7.470187 | 7.533345 | 14.474409 | 6.681065 | |
| Yellow | 2.648274 | 6.647290 | 9.215618 | 1.566533 | 6.895126 | 7.097645 | 8.779965 | 8.096916 | 15.449758 | |
| Orange | 3.549248 | 5.714639 | 9.266497 | 14.188935 | 6.878133 | 5.526465 | 7.916631 | 15.065882 | 4.996939 |
ซึ่ง PCA นั้นทำการลดมิติของข้อมูลลง โดยรวมข้อมูลอื่นๆ เข้าหาข้อมูลที่มีความสำคัญมากที่สุด นั่นคือ ข้อมูลที่มีความแปรปรวนสูงที่สุด ยกตัวอย่างข้อมูลสองมิติชุดหนึ่ง
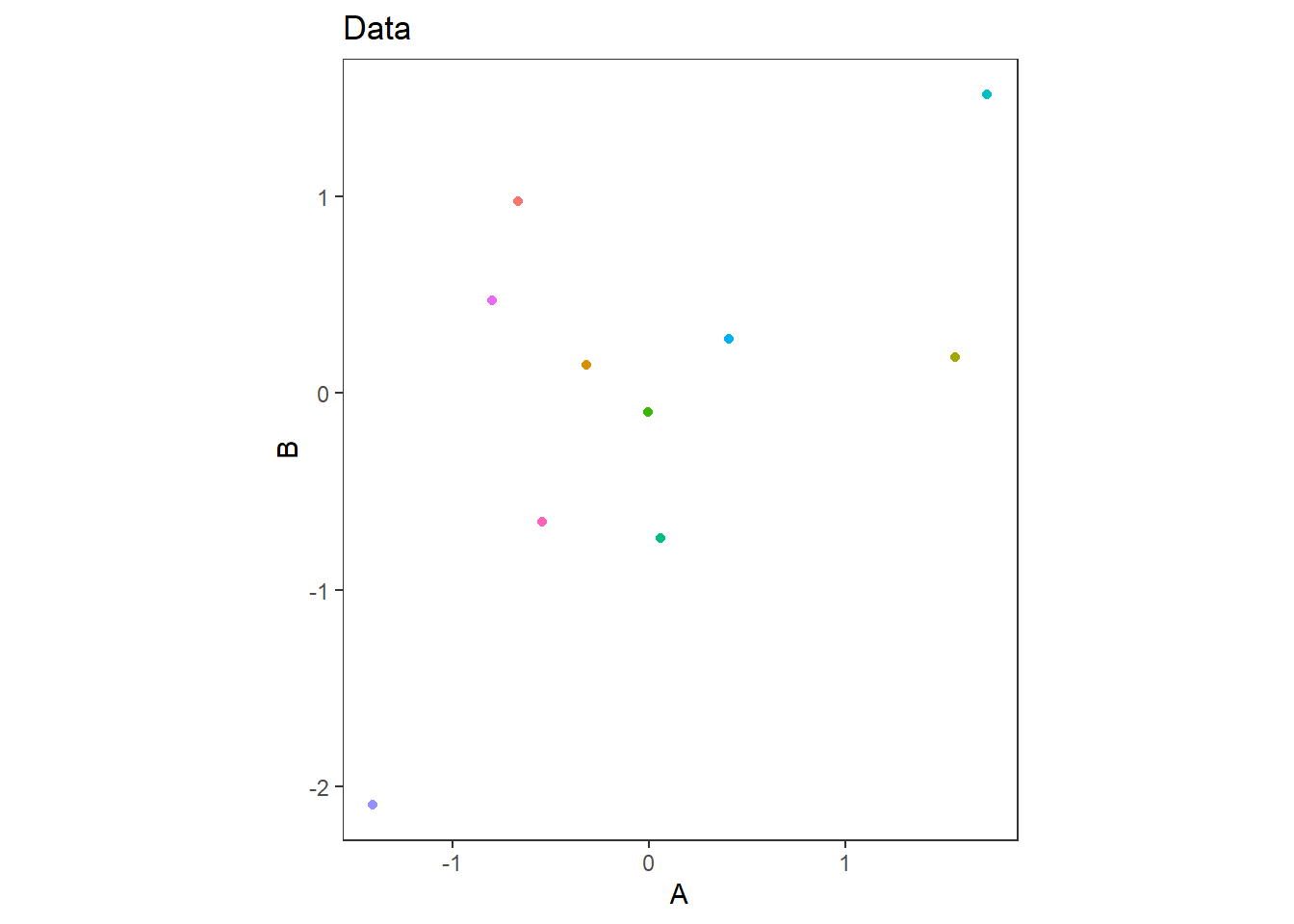
ในการวิเคราะห์ PCA ข้อมูลจะถูกลดมิติลงโดยการพยายามดึงเข้าสู่เส้นที่สามารถอธิบายความแปรปรวนได้ดีที่สุด ซึ่งก็คือ Principal component
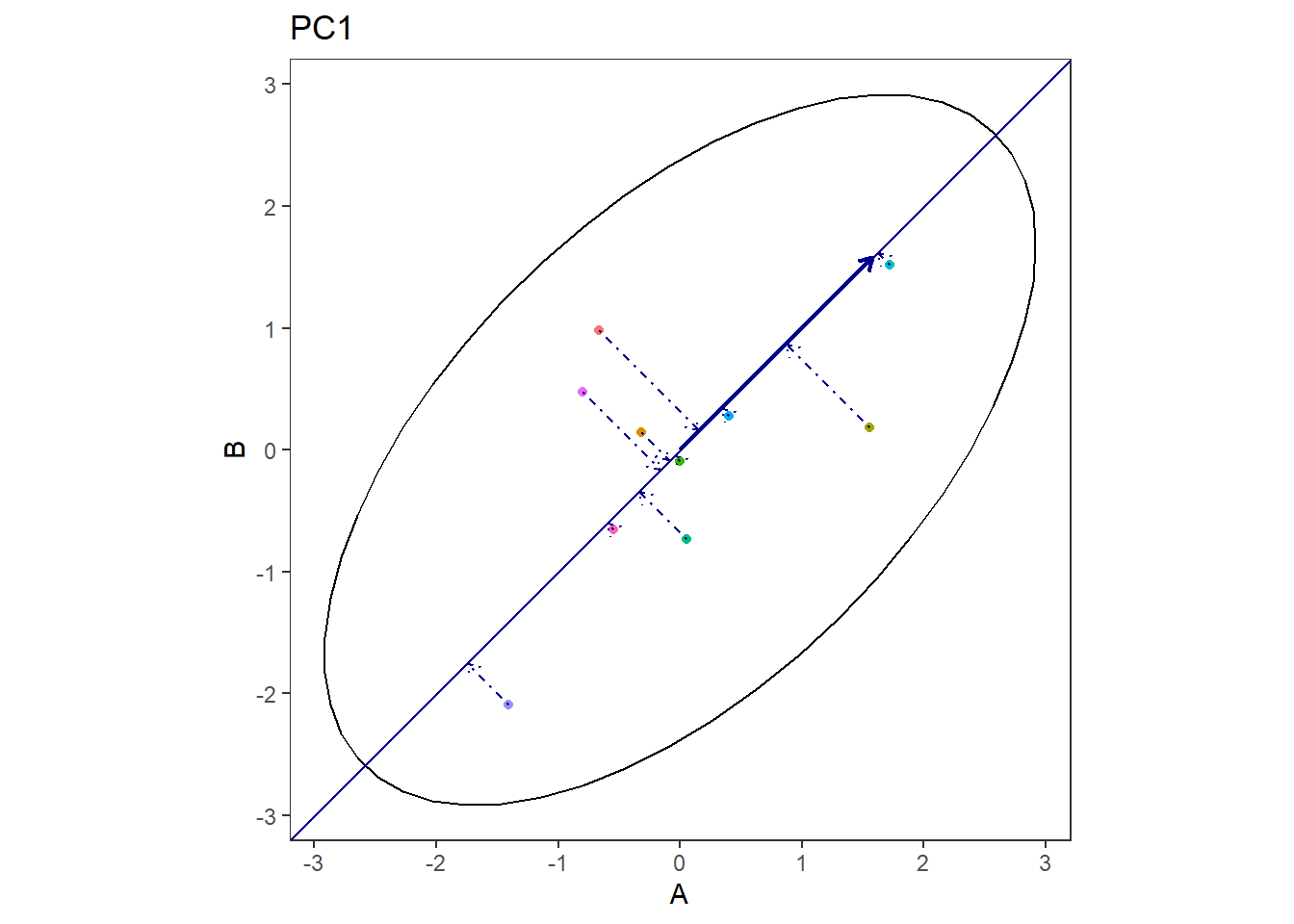
สังเกตว่า เส้นแนวเฉียงนั้น เป็นเส้นที่ข้อมูลแต่ละจุดห่างกันมากที่สุดใน 1 มิติ ซึ่งก็คือเส้นที่สามารถอธิบายความแปรปรวนได้ดีที่สุดนั่นเอง
ส่วนเส้นต่อมาที่ตั้งฉาก คือเส้นที่อธิบายความแปรปรวนได้ดีที่สุดเป็นลำดับสอง
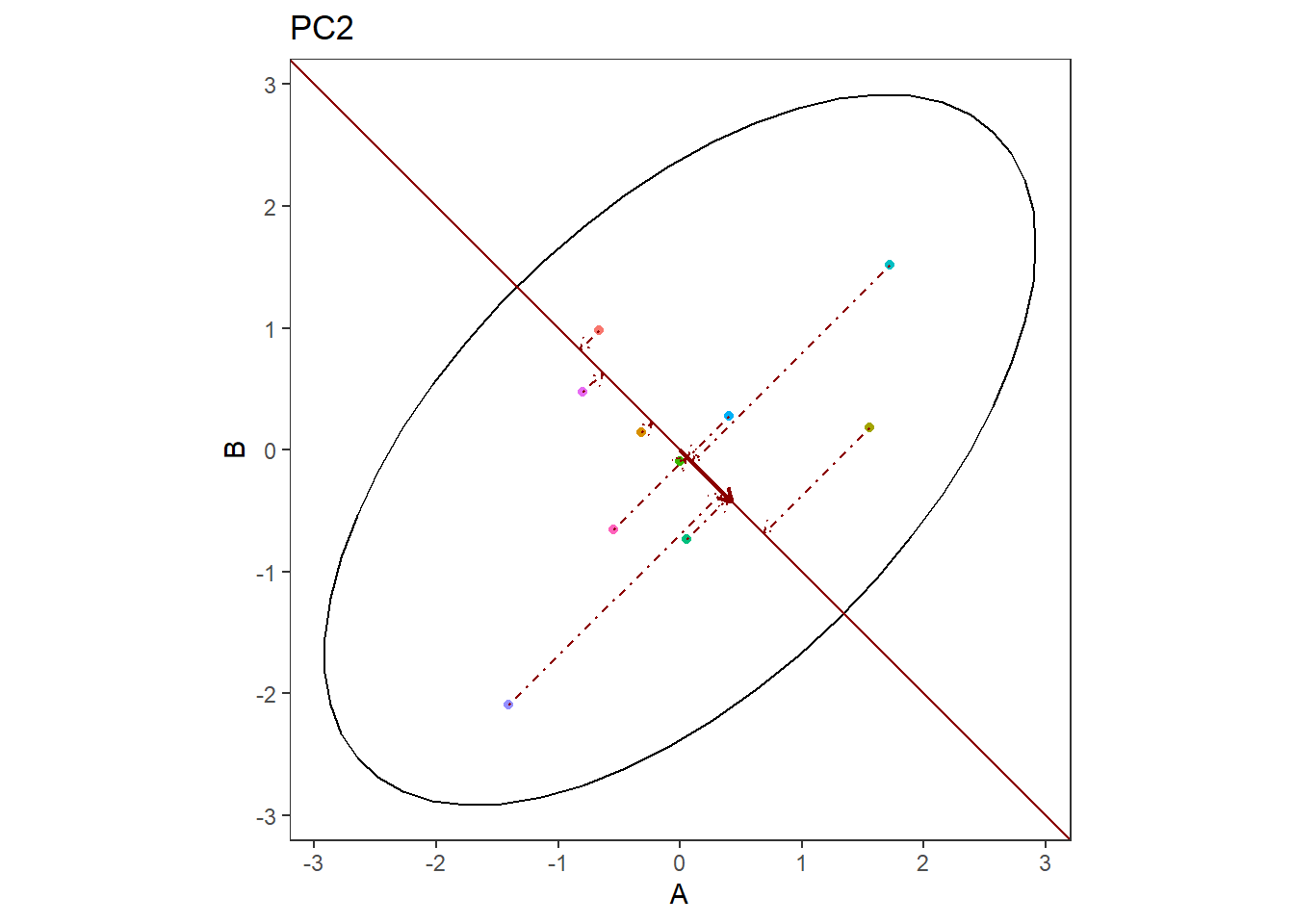
เมื่อนำข้อมูลทั้งหมดมาสร้างเป็นกราฟใหม่ จะได้ข้อมูลที่ยังลงเหลือความแปรปรวนที่มากที่สุดไว้ทั้งสองมิติ
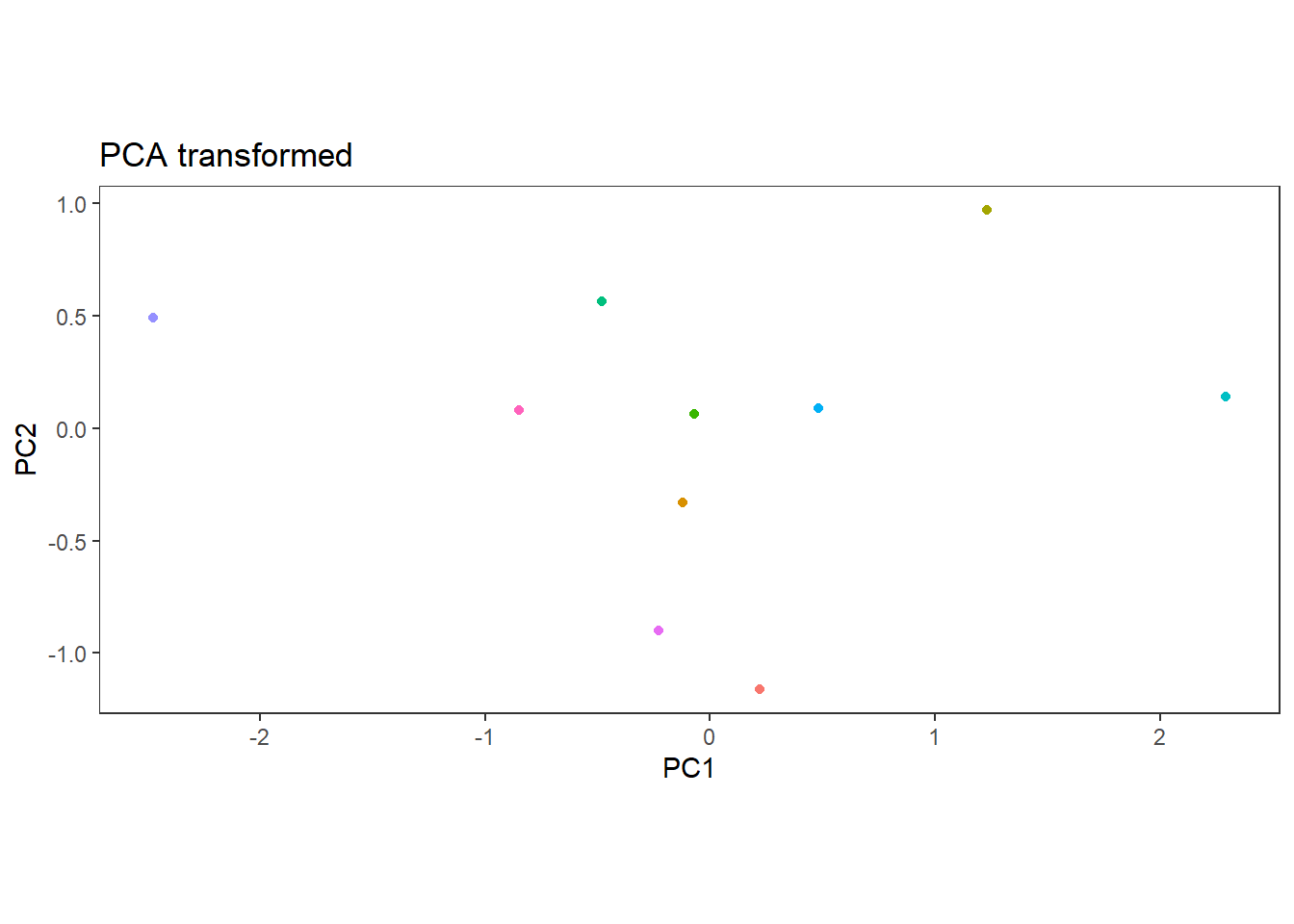
การลดมิติของข้อมูลนี้ อาจจะยังไม่เห็นผลนัก เนื่องจากข้อมูลมีแค่สองมิติเท่านั้น แต่ในสถานการณ์ซึ่งมีข้อมูลสูงมาก โดยเฉพาะเมื่อมี Feature มากกว่า Observations นั้น การลดข้อมูลจะมีประสิทธิภาพมาก โดยเฉพาะข้อมูลประเภท High-throughput (จำนวนยีนมากกว่าตัวอย่าง)
15.2 Manually calculate PCA
ต่อไปจะแสดงวิธีการคำนวณ PCA เพื่อให้เข้าใจลำดับของการวิเคราะห์
set.seed(123)
data <- matrix(c(
rnorm(10, mean = 20, sd = 2),
rnorm(10, mean = 1, sd = 2),
rnorm(10, mean = 4, sd = 5)),
ncol = 3
)
colnames(data) <- c("A", "B", "C")
data## A B C
## [1,] 18.87905 3.44816359 -1.3391185
## [2,] 19.53965 1.71962765 2.9101254
## [3,] 23.11742 1.80154290 -1.1300222
## [4,] 20.14102 1.22136543 0.3555439
## [5,] 20.25858 -0.11168227 0.8748037
## [6,] 23.43013 4.57382627 -4.4334666
## [7,] 20.92183 1.99570096 8.1889352
## [8,] 17.46988 -2.93323431 4.7668656
## [9,] 18.62629 2.40271180 -1.6906847
## [10,] 19.10868 0.05441718 10.2690746เริ่มต้นด้วยการ Normalize ข้อมูลให้อยู่ในรูป Mean-centering
## A B C
## [1,] -0.665875352 0.97821584 -0.6910813
## [2,] -0.319572479 0.14564661 0.2219402
## [3,] 1.555994430 0.18510203 -0.6461534
## [4,] -0.004316756 -0.09434713 -0.3269546
## [5,] 0.057310762 -0.73642490 -0.2153829
## [6,] 1.719927421 1.52040423 -1.3559540
## [7,] 0.405008410 0.27862049 1.3561812
## [8,] -1.404601888 -2.09545793 0.6208920
## [9,] -0.798376211 0.47466195 -0.7666212
## [10,] -0.545498338 -0.65642119 1.8031341scaled_data <- scale(data, center = TRUE, scale = TRUE) # Same
scaled_data## A B C
## [1,] -0.665875352 0.97821584 -0.6910813
## [2,] -0.319572479 0.14564661 0.2219402
## [3,] 1.555994430 0.18510203 -0.6461534
## [4,] -0.004316756 -0.09434713 -0.3269546
## [5,] 0.057310762 -0.73642490 -0.2153829
## [6,] 1.719927421 1.52040423 -1.3559540
## [7,] 0.405008410 0.27862049 1.3561812
## [8,] -1.404601888 -2.09545793 0.6208920
## [9,] -0.798376211 0.47466195 -0.7666212
## [10,] -0.545498338 -0.65642119 1.8031341
## attr(,"scaled:center")
## A B C
## 20.149251 1.417244 1.877206
## attr(,"scaled:scale")
## A B C
## 1.907568 2.076147 4.654046หลังจากนั้นเราจะทำการคำนวณ Covariance matrix ในที่นี้จะใช้แบบ Pearson’s
Note: ถ้ากลับไปพิจารณาสูตร Covariance Pearson’s แล้ว จะได้ว่า
cov=1n−1(ATA)
โดย n คือ จำนวนแถวของ Matrix A
cov_matrix <- t(scaled_data) %*% scaled_data / (nrow(scaled_data) - 1) # matrix multiplication
cov_matrix## A B C
## A 1.0000000 0.5776151 -0.4059593
## B 0.5776151 1.0000000 -0.5673487
## C -0.4059593 -0.5673487 1.0000000cov_matrix <- cov(scaled_data)
cov_matrix # same## A B C
## A 1.0000000 0.5776151 -0.4059593
## B 0.5776151 1.0000000 -0.5673487
## C -0.4059593 -0.5673487 1.0000000หลังจากนั้นเราทำการแยกทิศทางของความแปรปรวนออกมาโดยวิธี Eigendecomposition ซึ่งสำหรับ PCA แล้ว ซึ่ง Eigenvector นั้นบ่งบอกถึงทิศทางของความแปรปรวนของข้อมูลที่ตั้งฉากกัน โดยมีขนาดของการกระจายตัวของข้อมูล คือ Eigenvalue
eigen_info <- eigen(cov_matrix)
eigen_info## eigen() decomposition
## $values
## [1] 2.0376621 0.5941719 0.3681659
##
## $vectors
## [,1] [,2] [,3]
## [1,] -0.5596713 0.69413342 -0.4527105
## [2,] -0.6151534 0.01806984 0.7882003
## [3,] 0.5552966 0.71961954 0.4168854โดย vectors คือ Eigenvector หรือ ทิศทางของความแปรปรวน (เรียงตามคอลัมน์) ส่วน values คือ Eigenvalues หรือ ขนาดของความแปรปรวน
Note: เราสามารถคำนวณ Eigenvalue และ Eigenvector กลับไปเป็น Covariance matrix
A=VQV−1
eigen_back <-
eigen_info$vectors %*% diag(eigen_info$values) %*% solve(eigen_info$vectors)
round(sum(eigen_back - cov_matrix), 10) # Diff nearly 0 due to algorithm## [1] 0ต่อไปจะต้องเรียง Eigenvector ตามขนาดของ Eigenvalues จากน้อยไปมาก ซึ่งนั้นก็คือ Principal component ของข้อมูล (PC1, PC2, PC3, ….) นั่นหมายความว่า nPC≤nObs เสมอ
sorted_eigenvalues <- eigen_info$values
sorted_eigenvectors <- eigen_info$vectors[, order(-sorted_eigenvalues)] # Decreasing ordernum_components <- 3 # Adjust this as needed
selected_eigenvectors <- sorted_eigenvectors[, 1:num_components]สุดท้ายเมื่อทำการคูณข้อมูลเดิมกลับไปด้วย Eigenvectors ที่เรียงแล้ว จะได้ ข้อมูลของความแปรปรวนข้อมูลแต่ละคอลัมน์ในแต่ละ PC
pca_result <- scaled_data %*% selected_eigenvectors
colnames(pca_result) <- paste0("PC", 1:ncol(selected_eigenvectors))
pca_result## PC1 PC2 PC3
## [1,] -0.6128366 -0.94184573 0.7843772
## [2,] 0.2125032 -0.05948164 0.3519961
## [3,] -1.3435184 0.61842786 -0.8278895
## [4,] -0.1211029 -0.23998416 -0.2087128
## [5,] 0.3013377 -0.12851951 -0.6961855
## [6,] -2.6508325 0.24556160 -0.1455235
## [7,] 0.3550169 1.26209896 0.6016293
## [8,] 2.4199227 -0.56603968 -0.7569218
## [9,] -0.2708638 -1.09727813 0.4159689
## [10,] 1.7103737 0.90706045 0.481261715.3 PCA by R function
ท่านไม่จำเป็นต้องคำนวณ PCA ตามทั้งหมดที่กล่าวมาขั้นต้น เนื่องจาก R ได้มีฟังก์ชันสำหรับวิเคราะห์ PCA ไว้ให้แล้ว ในที่นี้จะลองสร้างสถานการณ์ที่ Features > Observations
set.seed(123)
data <- matrix(
sapply(1:10, \(x) rnorm(5, mean = sample(1:20, 1), sd = sample(1:10, 1))),
ncol = 10
)
colnames(data) <- LETTERS[1:10]
as.data.frame(data)A <dbl> | B <dbl> | C <dbl> | D <dbl> | E <dbl> | F <dbl> | G <dbl> | H <dbl> | I <dbl> | J <dbl> |
|---|---|---|---|---|---|---|---|---|---|
| 18.570620 | 10.070553 | 21.652869 | 2.400106 | 17.284334 | 8.769588 | 0.8768292 | -2.594688 | 15.131765 | 6.000378 |
| 9.931333 | 6.511437 | 17.429859 | 9.402037 | 9.838178 | 5.690441 | 28.3516477 | 4.144220 | -9.390022 | 3.944274 |
| 18.718488 | 12.585010 | 9.498826 | 17.494274 | 3.344357 | 4.470187 | 20.6636960 | 1.839351 | 7.676910 | 3.784626 |
| 14.673102 | 6.647290 | 2.940560 | 8.982245 | 14.056131 | 4.097645 | 2.0151313 | 12.449363 | 3.990834 | 7.910586 |
| 14.648274 | 5.714639 | 3.398471 | 1.841186 | 13.903201 | 2.526465 | 7.7769213 | 10.141964 | 4.727533 | 8.344629 |
## List of 5
## $ sdev : num [1:5] 2.03 1.88 1.45 4.94e-01 2.83e-16
## $ rotation: num [1:10, 1:5] -0.196 -0.424 -0.336 -0.274 0.267 ...
## ..- attr(*, "dimnames")=List of 2
## .. ..$ : chr [1:10] "A" "B" "C" "D" ...
## .. ..$ : chr [1:5] "PC1" "PC2" "PC3" "PC4" ...
## $ center : Named num [1:10] 15.31 8.31 10.98 8.02 11.69 ...
## ..- attr(*, "names")= chr [1:10] "A" "B" "C" "D" ...
## $ scale : Named num [1:10] 3.61 2.92 8.36 6.37 5.36 ...
## ..- attr(*, "names")= chr [1:10] "A" "B" "C" "D" ...
## $ x : num [1:5, 1:5] -1.242 -0.599 -2.375 1.895 2.322 ...
## ..- attr(*, "dimnames")=List of 2
## .. ..$ : NULL
## .. ..$ : chr [1:5] "PC1" "PC2" "PC3" "PC4" ...
## - attr(*, "class")= chr "prcomp"rotationคือ Eigenvectors ที่คำนวณจากeigensdevคือ รากที่สองของ Eigenvalues ที่คำนวณจากeigenxคือ ข้อมูลสุดท้ายที่ถูกลดมิติแล้ว
สังเกตว่าสุดท้าย PC จะไม่เกินจำนวน Observations = 5
pc$x## PC1 PC2 PC3 PC4 PC5
## [1,] -1.2422037 -2.971150049 -0.8164442 -0.009920526 3.701466e-16
## [2,] -0.5994732 2.075884300 -1.9908362 -0.039263516 4.440892e-16
## [3,] -2.3753407 0.963616858 1.7962551 0.097225061 -2.220446e-16
## [4,] 1.8951608 -0.003007043 0.6758462 -0.718606015 3.330669e-16
## [5,] 2.3218568 -0.065344065 0.3351791 0.670564995 2.220446e-1615.3.1 Biplot
คือการพล็อตกราฟเพื่อบ่งบอก ว่า แต่ละ Features ดั้งเดิมนั้น มีผลต่อ PC มากน้อยเพียงใด
biplot(pc)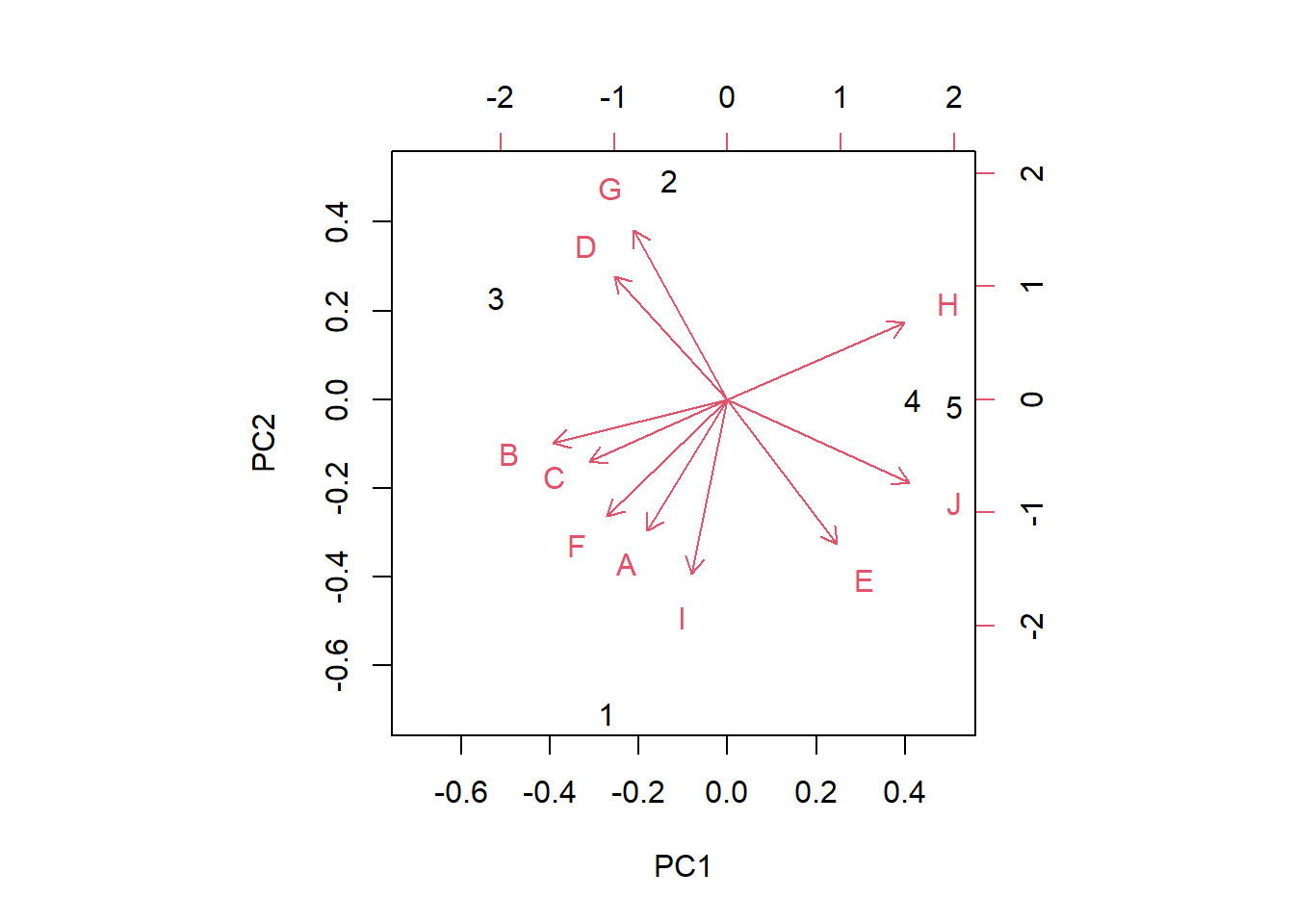
โดยทิศทางที่ชี้ไปนั้นคืออิทธิพลของ Features ดั้งเดิมที่ประกอบเป็น PC
15.3.2 Screeplot
เป็นการพล็อตเพื่อสำรวจว่าแต่ละ PC นั้นมีอิทธิผลจากข้อมูลมากเท่าใด ซึ่งก็คือการดูขนาดของ Eigenvalues หรือ ความแปรปรวนของ PC นั้นๆ
ggplot(data = NULL, aes(x = 1:5)) +
geom_col(aes(y = pc$sdev^2),fill = "skyblue", col = "black") +
geom_line(aes(y = cumsum(pc$sdev^2))) +
geom_text(aes(y = pc$sdev^2,
label = paste0(round(pc$sdev^2,2),"%"), vjust = -2)) +
labs(x = "PC", y = "Variance (%)") +
theme_bw()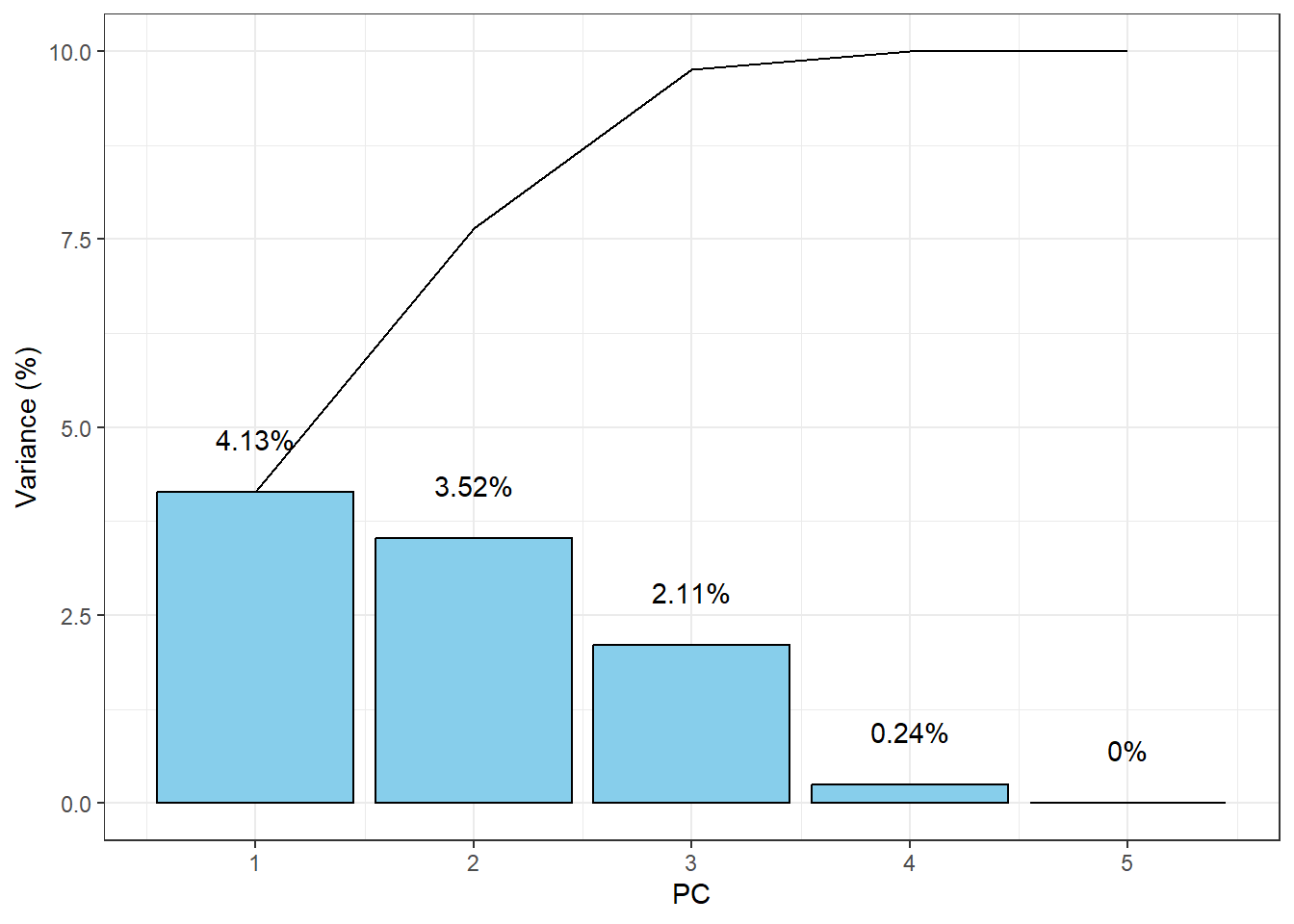
หรือท่านอาจจะเรียก screeplot()
screeplot(pc)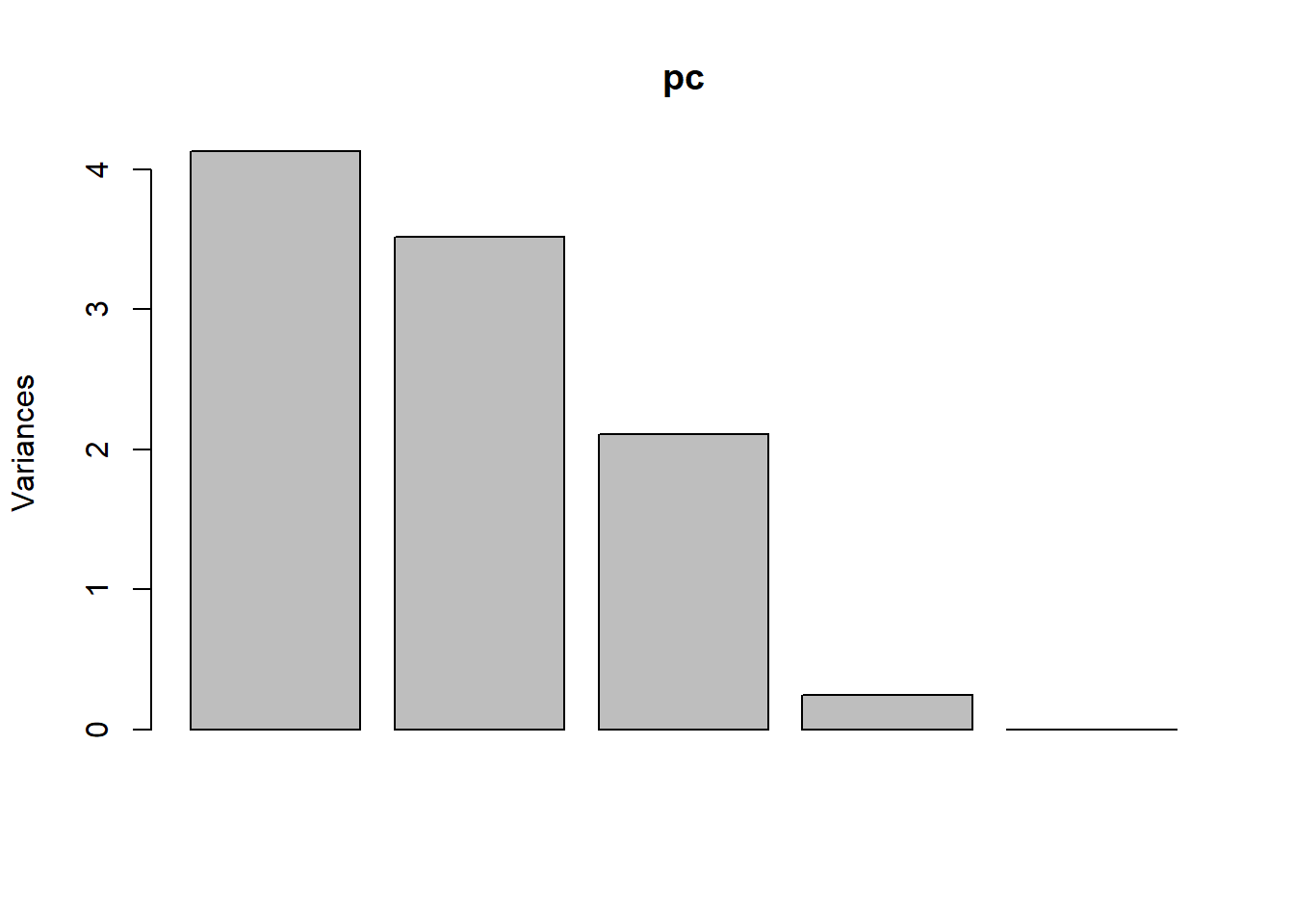
สังเกตว่า PC จะเรียงจากมากไปน้อยเสมอ ตามขนาดของ Eigenvalue