10 Parametric test
คือ การวิเคราะห์ทางสถิติที่เราทราบลักษณะการกระจายตัวของข้อมูลอย่างชัดเจน แต่มีข้อดีคือการเปรียบเทียบข้อมูลจะมีความแม่นยำสูง
10.1 \(t\)-test
คือ การคำนวณทางสถิติที่เปรียบเทียบค่าเฉลี่ย (Mean) และความผันผวนของการวัด (Standard Error; SE) ระหว่างสองกลุ่ม แบ่งเป็น
10.1.1 One sample \(t\)-test
เป็นการเปรียบเทียบความแตกต่างของค่าเฉลี่ยระหว่างกลุ่มตัวอย่าง (Sample) กับประชากร (Population)
\[ t = \frac{\bar{x} - \mu_{0}}{SE} = \frac{\bar{x} - \mu_{0}}{s/\sqrt{n}} \]
เพื่อให้เห็นภาพของความต่างในการทดสอบนี้ เราจะทำการสร้างกราฟเปรียบว่ากลุ่มตัวอย่างนั้น มีค่าเฉลี่ยต่างจากประชากร ที่ ค่าเฉลี่ย = 10 และ ค่าเฉลี่ย = 30 หรือไม่
set.seed(123)
norm_10_pop <- rnorm(1000, mean = 10, sd = 4) # สร้างประชากร mean = 10, sd = 4
norm_10_sample <- sample(norm_10_pop, 100) # สุ่มตัวอย่างมาจากประชากร 100 ราย
norm_30_pop <- rnorm(1000, mean = 30, sd = 4)
norm_30_sample <- sample(norm_30_pop, 100)
norm_pop_df <- data.frame(Pop_10 = norm_10_pop, Pop_30 = norm_30_pop) |>
pivot_longer(everything(), names_to = "Pop", values_to = "Values")
norm_sample_df <- data.frame(Samp_10 = norm_10_sample, Samp_30 = norm_30_sample) |> pivot_longer(everything(), names_to = "Samp", values_to = "Values")
ggplot(norm_pop_df, aes(x = Values, fill = Pop)) +
geom_density(aes(y = after_stat(count)),color = "black", alpha = 0.5) +
geom_density(data = norm_sample_df,
aes(x = Values, y = after_stat(count), fill = Samp),
color = "black", alpha = 0.7) + theme_bw()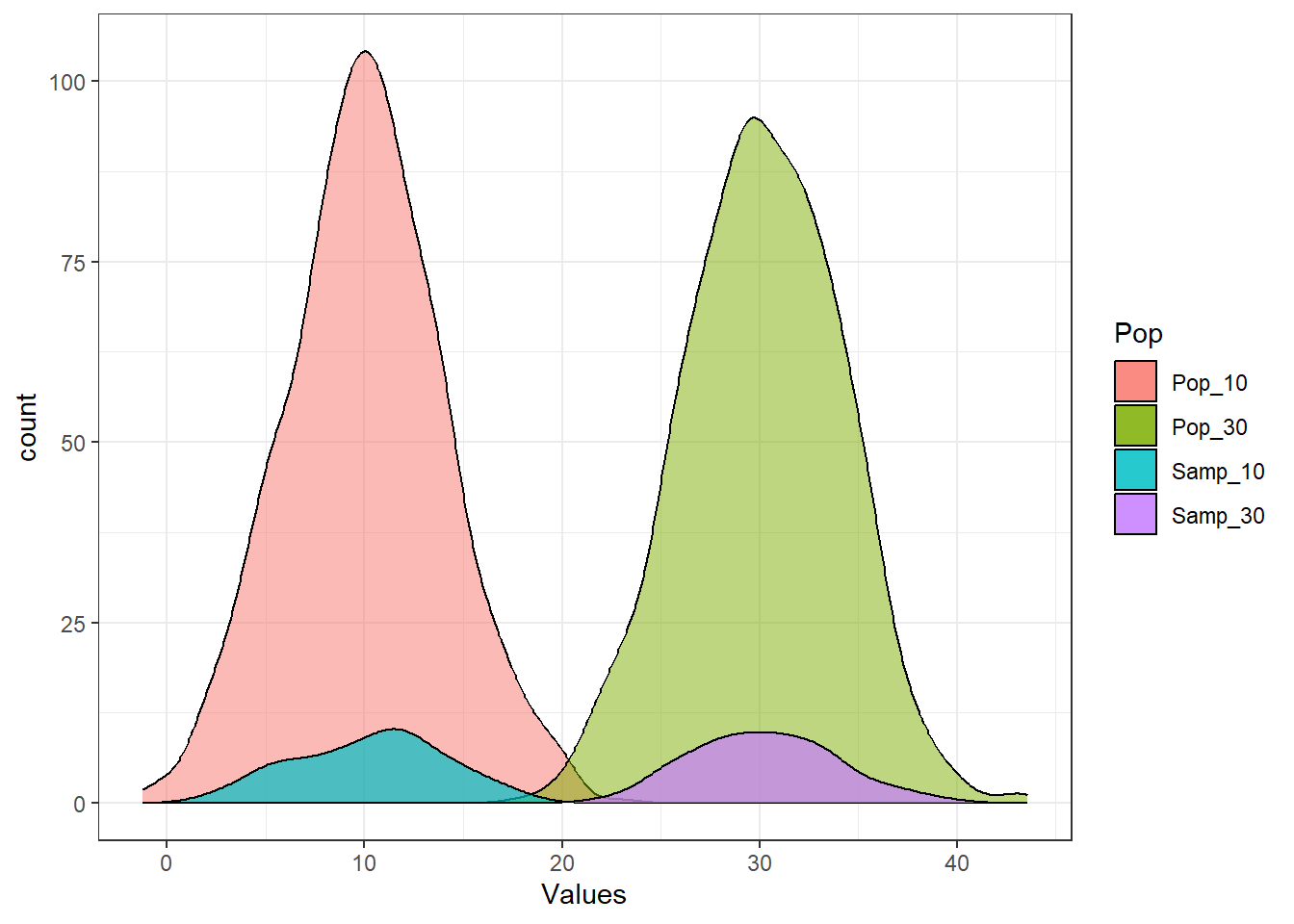
จะเห็นว่าเมื่อดูลักษณะการกระจายตัวของข้อมูลแล้ว Samp_10 ที่มี ค่าเฉลี่ย = 10 นั้น ไม่ต่างจากประชากรที่มี ค่าเฉลี่ย = 10 แต่ดูแตกต่างอย่างเห็นได้ชัดกับประชากรที่มี mean = 30 t.test จะช่วยตัดสินว่าค่าที่ได้นั้นแตกต่างกันจริงหรือไม่ โดยสมมติฐานที่ตั้งคือ
Case 1: ประชากรมีค่าเฉลี่ย = 10
\(H_{0}\): ค่าเฉลี่ยของกลุ่มตัวอย่างนั้น ไม่ต่างจากค่าเฉลี่ยของประชากร = 10
\(H_{a}\): ค่าเฉลี่ยของกลุ่มตัวอย่างนั้น ต่างจากค่าเฉลี่ยของประชากร = 10
t.test(norm_10_sample, mu = 10) # p > 0.05##
## One Sample t-test
##
## data: norm_10_sample
## t = 0.73547, df = 99, p-value = 0.4638
## alternative hypothesis: true mean is not equal to 10
## 95 percent confidence interval:
## 9.533936 11.015052
## sample estimates:
## mean of x
## 10.27449Case 2: ประชากรมีค่าเฉลี่ย = 40
\(H_{0}\): ค่าเฉลี่ยของกลุ่มตัวอย่างนั้น ไม่ต่างจากค่าเฉลี่ยของประชากร = 40
\(H_{a}\): ค่าเฉลี่ยของกลุ่มตัวอย่างนั้น ต่างจากค่าเฉลี่ยของประชากร = 40
t.test(norm_10_sample, mu = 30) # p <= 0.05##
## One Sample t-test
##
## data: norm_10_sample
## t = -52.852, df = 99, p-value < 2.2e-16
## alternative hypothesis: true mean is not equal to 30
## 95 percent confidence interval:
## 9.533936 11.015052
## sample estimates:
## mean of x
## 10.27449จะเห็นว่าต่างจากประชากรที่ค่าเฉลี่ย = 30 อย่างมีนัยสำคัญ (\(p\) = \(2.2 \times 10^{-16}\) < Critical point = 0.05)
พิจารณาที่มาของ \(p\)-value นั้นมีที่มาจากสูตรขั้นต้น
hypothesized_mean <- 30
mean_sample <- mean(norm_10_sample)
sd_sample <- sd(norm_10_sample)
t <- (mean_sample - hypothesized_mean)/(sd_sample/sqrt(length(norm_10_sample)))
t## [1] -52.85159
2*pt(t, df = 99) # p-value## [1] 2.306252e-74จะเห็นว่าค่า \(t\) นั้นเท่ากับ ค่าที่ได้จาก t.test ขั้นต้น เมื่อสร้าง \(t\)-distribution ที่มี \(H_{0}\) คือ ค่าเฉลี่ย = 0 แล้วจะพบว่าค่านี้มากกว่า Critical value
10.1.2 Independent t-test
เป็นการเปรียบเทียบค่าเฉลี่ยระหว่าง ตัวอย่างสองกลุ่มที่ไม่เกี่ยวเนื่องกัน
\[ t = \frac{\bar{x_{1}}-\bar{x_{2}}-\mu_{0}}{\sqrt{\frac{s^{2}_1}{n_{1}}+\frac{s^{2}_2}{n_{2}}}} \]
\[ s_{p} = \sqrt{\frac{(n_{1}-1)s^{2}_{1}+(n_{2}-1)s^{2}_{2}}{{n_{1}} + n_{2} - 2}} \]
กลับไปดูภาพขั้นต้น ครั้งนี้จะเทียบระหว่างกลุ่มตัวอย่างสองกลุ่ม คือ norm_10_sample และ norm_30_sample ว่ามี ค่าเฉลี่ยที่แตกต่างกันอย่างมีนัยสำคัญหรือไม่
\(H_{0}\): ค่าเฉลี่ยของ
norm_10_sampleและnorm_30_sampleนั้นไม่แตกต่างกัน (\(\bar{x_{1}} - \bar{x_{2}} = 0\))\(H_{a}\): ค่าเฉลี่ยของ
norm_10_sampleและnorm_30_sampleนั้นแตกต่างกัน (\(\bar{x_{1}} - \bar{x_{2}} \neq 0\))
t.test(norm_10_sample, norm_30_sample, var.equal = TRUE)##
## Two Sample t-test
##
## data: norm_10_sample and norm_30_sample
## t = -38.469, df = 198, p-value < 2.2e-16
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -20.93623 -18.89440
## sample estimates:
## mean of x mean of y
## 10.27449 30.18981สรุปได้ว่าค่าเฉลี่ยของกลุ่มตัวอย่างทั้งสองกลุ่มนั้นแตกต่างกันอย่างมีนัยสำคัญ
ลองคำนวณเอง
ind_t_df <- data.frame(ten = norm_10_sample, thirty = norm_30_sample) |>
summarize(across(c(everything()), .fns =
list(mean = mean, sd = sd)))
ind_t_df## [1] 0.5176993
ind_t <- (ind_t_df$ten_mean - ind_t_df$thirty_mean - 0)/ind_t_se
ind_t## [1] -38.46889## [1] 7.928494e-94Note: บางครั้งข้อมูลอาจจะมีความแปรปรวนไม่เท่ากัน ในที่นี้มักจะใช้ Welch’s \(t\)-test โดย t.test(…, var.equal = FALSE) โดยสมการนี้จะทำการปรับความแปรปรวนให้ด้วย
10.1.3 Paired t-test
เป็นการเปรียบเทียบค่าเฉลี่ยระหว่างตัวอย่างสองกลุ่มที่เกี่ยวเนื่องกัน (ก่อน-หลัง แม่-ลูก เป็นต้น)
\[ t = \frac{\bar{x}_{d}-\mu_{0}}{s_{x_{d}}/\sqrt{n}} \]
\[ x_{di} = x_{1_{i}}-x_{2_{i}} \]
\[ s_{x_{d}} = \sqrt{\frac{\sum_{d=1}^{n}(x_{d}-\bar{x}_{d})^{2}}{(n-1)}} \]
t.test(norm_10_sample, norm_30_sample, paired = TRUE, var.equal = TRUE)##
## Paired t-test
##
## data: norm_10_sample and norm_30_sample
## t = -37.802, df = 99, p-value < 2.2e-16
## alternative hypothesis: true mean difference is not equal to 0
## 95 percent confidence interval:
## -20.96067 -18.86996
## sample estimates:
## mean difference
## -19.91532จะเห็นว่ามีความแตกต่างในส่วนของ \(df\) (Degree of freedom) ซึ่งเกิดจากการจับคู่หาความต่าง 100 ครั้ง เนื่องจากค่าที่เปรียบเทียบนั้นอยู่ในตัวอย่างเดียวกัน (แม่ลูก คู่ที่ 1 แม่ลูกคู่ที่ 2 … เป็นต้น) เมื่อเปรียบเทียบกับ Independent \(t\)-test เนื่องจากเป็นการหา mean ในกลุ่มของตัวเอง 2 ครั้ง และมาเทียบความต่างกัน
ลองคำนวณเอง
paired_t_df <- data.frame(ten = norm_10_sample, thirty = norm_30_sample) |>
mutate(diff = ten -thirty) |>
mutate(mean_diff = mean(diff)) |>
mutate(ss_mean_diff = (diff-mean_diff)^2)
paired_t_df## [1] 5.268367## [1] -37.80168## [1] 1.223407e-60ดังนั้น การเลือก Paired vs Independent นั้นมีความสำคัญ ขึ้นอยู่กับโจทย์การศึกษาของท่านด้วย
10.2 Analysis of Variance (ANOVA)
คือ การคำนวณทางสถิติที่เปรียบเทียบค่าเฉลี่ยและความแปรปรวนระหว่างสองกลุ่มขึ้นไป โดยมีสมมติฐาน คือ
\(H_{0}\): ค่าเฉลี่ยของทุกกลุ่มเท่ากัน (\(\bar{x_{1}} = \bar{x_{2}} = \bar{x_{3}} = … = \bar{x_{n}}\))
\(H_{a}\): ค่าเฉลี่ยของกลุ่มใดกลุ่มหนึ่งต่างจากกลุ่มอื่น
การวิเคราะห์ ANOVA นั้นมีหลายแบบ
One-way ANOVA เป็นการหาความแตกต่างของ 1 ตัวแปร
Two-way ANOVA เป็นการหาความแตกต่างของ 2 ตัวแปร
MANOVA เป็นการหาความแตกต่างที่มากกว่า 2 ตัวแปร
Nested ANOVA เป็นการหาความแตกต่างที่ใน 1 ตัวแปรนั้น มีตัวแปรย่อยอีก
ณ ที่นี้จะกล่าวถึงแค่ one ANOVA ซึ่งมีความซับซ้อนน้อย และนิยมใช้มากที่สุด โดยหลักการโดยง่ายของ ANOVA คือ การเปรียบเทียบความต่างของ ค่าเฉลี่ยทั้งกลุ่ม (Global mean) เปรียบเทียบกับ ผลรวมของค่าเฉลี่ยแต่ละกลุ่ม (Between group mean)
การวิเคราะห์ทางสถิติของ ANOVA นั้นใช้ \(F\)-test ซึ่งเป็นการเปรียบเทียบระหว่าง ความแปรปรวนที่อธิบายได้ และความแปรปรวนที่อธิบายไม่ได้
\[F^{*} = \frac{\text{Explained variance}}{\text{Unexplained variance}} = \frac{\text{Between group variance}}{\text{Within groups variance}}\]
Between group variance คือ ความแปรปรวนของค่าเฉลี่ยแต่ละกลุ่มกับค่าเฉลี่ยทั้งหมด
Within group variance คือ ความแปรปรวนของข้อมูลในกลุ่มนั้น ซึ่งไม่สามารถอธิบายได้ภายใต้สมมติฐานงานวิจัยนั้น
อธิบายโดยใช้ตัวอย่าง iris ในที่นี้เราจะเปรียบเทียบตวามต่างของ Sepal.Width ในแต่ละ Species
## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 11.35 5.672 49.16 <2e-16 ***
## Residuals 147 16.96 0.115
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1อธิบายด้วยภาพ
library(granova)
granova.1w(iris$Sepal.Width, group = iris$Species)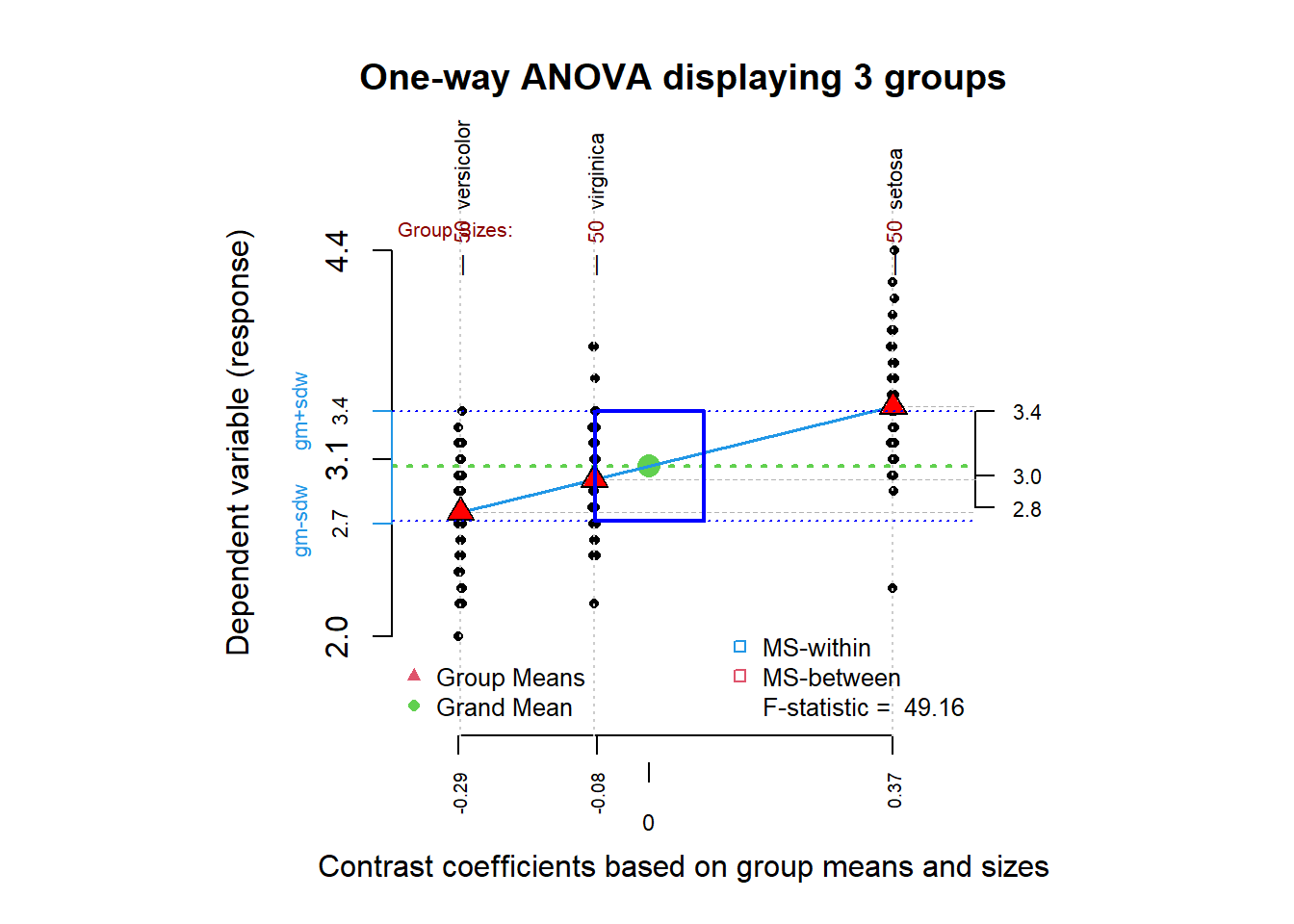
## $grandsum
## Grandmean df.bet df.with MS.bet MS.with F.stat F.prob
## 3.06 2.00 147.00 5.67 0.12 49.16 0.00
## SS.bet/SS.tot
## 0.40
##
## $stats
## Size Contrast Coef Wt'd Mean Mean Trim'd Mean Var. St. Dev.
## versicolor 50 -0.29 2.77 2.77 2.80 0.10 0.31
## virginica 50 -0.08 2.97 2.97 2.96 0.10 0.32
## setosa 50 0.37 3.43 3.43 3.41 0.14 0.38รูปสามเหลี่ยม คือ ค่าเฉลี่ยของ
Petal.Widthในดอกไม้แต่ละSpeciesจุดสีดำ คือ ค่า
Petal.Widthในดอกไม้แต่ละดอกจุดสีเขียว คือ Global mean (Grand mean) คือ ค่าเฉลี่ย
Petal.Widthเมื่อรวมดอกไม้ทุกSpecies
จุดประสงค์ของ F-test คือการเปรียบเทียบอัตราส่วนระหว่าง ความแปรปรวนของค่าเฉลี่ยแต่ละกลุ่มกับค่าเฉลี่ยทั้งหมด (mean ของความต่างจุดเขียวกับสามเหลี่ยม) ใกล้กันกับความแปรปรวนของข้อมูลในกลุ่มนั้น (mean ของความต่างระหว่างจุดดำกับกับสามเหลี่ยม) หรือไม่ (ratio ~ 1) ซึ่งค่า \(F\) นั้นจะถูกนำไปคิด \(p\)-value จาก \(F\)-distribution (หลักการเดียวกันกับ \(t\)-test)
สังเกตว่า ท่านไม่สามารถบอกได้ว่ากลุ่มไหนเป็นกลุ่มที่มีค่าเฉลี่ยที่แตกต่างจากทั้งกลุ่ม การที่จะทราบนั้นต้องทำ t.test เปรียบเทียบกันในแต่ละกลุ่ม \(3\choose2\) = 3 ครั้ง การค้นหากลุ่มที่มีความต่างหลัง ANOVA นี้ เรียกว่า Post-hoc test
pairwise.t.test(iris$Sepal.Width, iris$Species)##
## Pairwise comparisons using t tests with pooled SD
##
## data: iris$Sepal.Width and iris$Species
##
## setosa versicolor
## versicolor < 2e-16 -
## virginica 9.1e-10 0.0031
##
## P value adjustment method: holm\(p\)-value < 0.05 ทุกกลุ่ม หมายความว่า ทุกกลุ่มมีค่าเฉลี่ยของ Petal.Width ที่แตกต่างกัน
10.3 Correlation test
10.3.1 Pearson correlation
เป็นการทดสอบความสัมพันธ์เชิงเส้นของตัวแปรสองตัวแปร
\[ r_{xy} =\frac{cov(x,y)}{s(x) s(y)} \]
\[ cov(x,y) = \frac{\sum^{n}_{i=1}(x_{i}-\bar{x})(y_{i}-\bar{y})}{n-1} \]
\[ \therefore r_{xy} = \frac{\sum^{n}_{i=1}(x_{i}-\bar{x})(y_{i}-\bar{y})}{\sqrt{\sum^{n}_{i=1}(x_{i}-\bar{x})^{2}}\sqrt{\sum^{n}_{i=1}(y_{i}-\bar{y})^{2}}} \]
ggplot(iris, aes(x = Sepal.Length, y = Petal.Length)) +
geom_point(aes(col = Species)) +
geom_smooth(method = "lm", se = FALSE) +
theme_bw()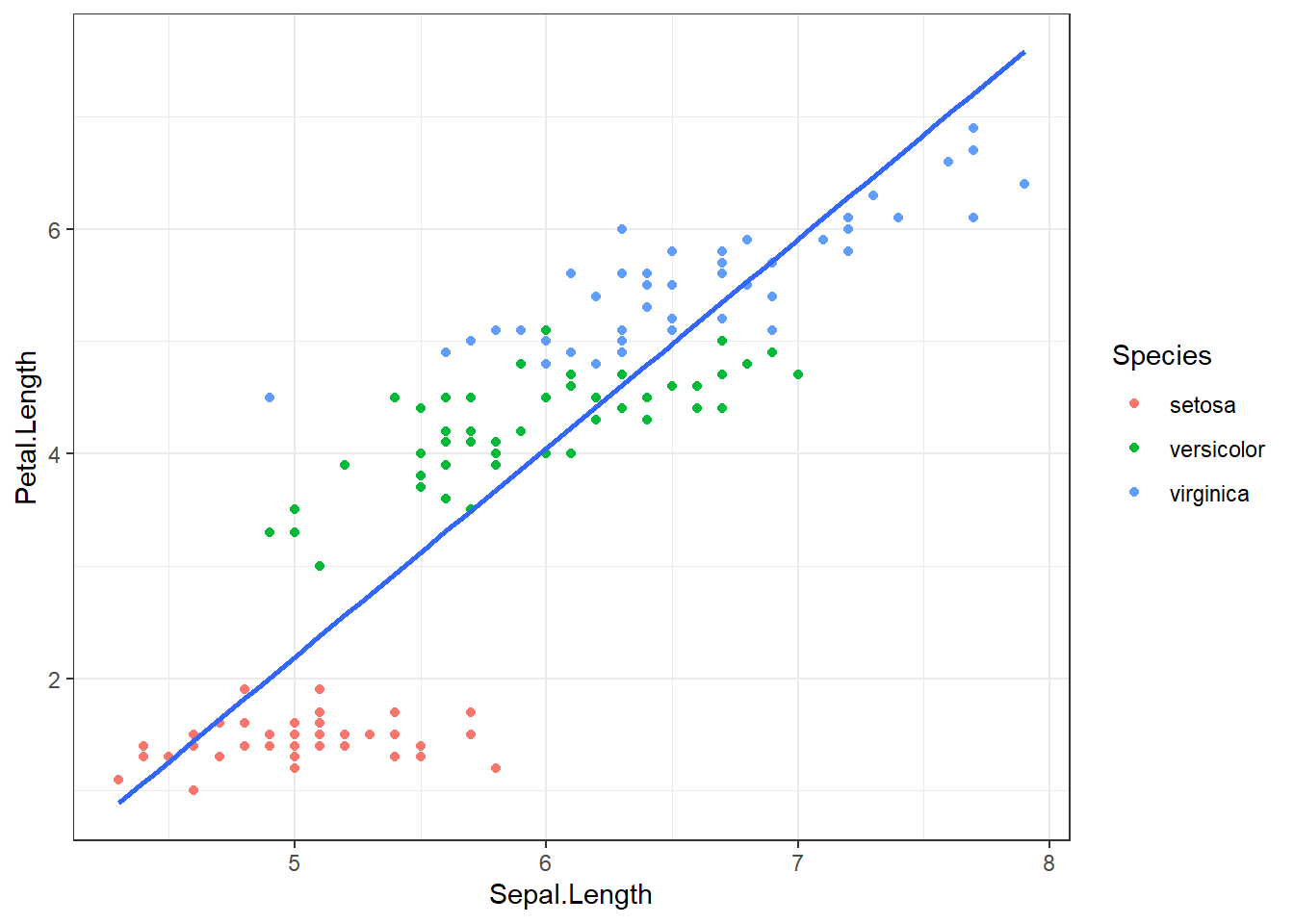
cor(iris$Sepal.Length, iris$Petal.Length)## [1] 0.8717538การแปลผลของ Correlation ขึ้นอยู่กับ
ระดับของความสัมพันธ์ = 0-1 ยิ่งเข้าใกล้ 1 ยิ่งสัมพันธ์กันมาก
ทิศทางของความสัมพันธ์ = + ไปทิศทางเดียวกัน - ไปทิศทางตรงข้ามกัน
ท่านสามารถสร้างตาราง Correlation และ \(p\)-value ได้ด้วย Package corrplot
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411
## Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259
## Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654
## Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000
iris_cor_p <- cor.mtest(iris[1:4])
iris_cor_p## $p
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.000000e+00 1.518983e-01 1.038667e-47 2.325498e-37
## Sepal.Width 1.518983e-01 0.000000e+00 4.513314e-08 4.073229e-06
## Petal.Length 1.038667e-47 4.513314e-08 0.000000e+00 4.675004e-86
## Petal.Width 2.325498e-37 4.073229e-06 4.675004e-86 0.000000e+00
##
## $lowCI
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 -0.2726932 0.8270363 0.7568971
## Sepal.Width -0.2726932 1.0000000 -0.5508771 -0.4972130
## Petal.Length 0.8270363 -0.5508771 1.0000000 0.9490525
## Petal.Width 0.7568971 -0.4972130 0.9490525 1.0000000
##
## $uppCI
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.00000000 0.04351158 0.9055080 0.8648361
## Sepal.Width 0.04351158 1.00000000 -0.2879499 -0.2186966
## Petal.Length 0.90550805 -0.28794993 1.0000000 0.9729853
## Petal.Width 0.86483606 -0.21869663 0.9729853 1.0000000
corrplot(iris_cor, method = "shade",type = "upper",order = "AOE",
p.mat = iris_cor_p$p, insig = "p-value")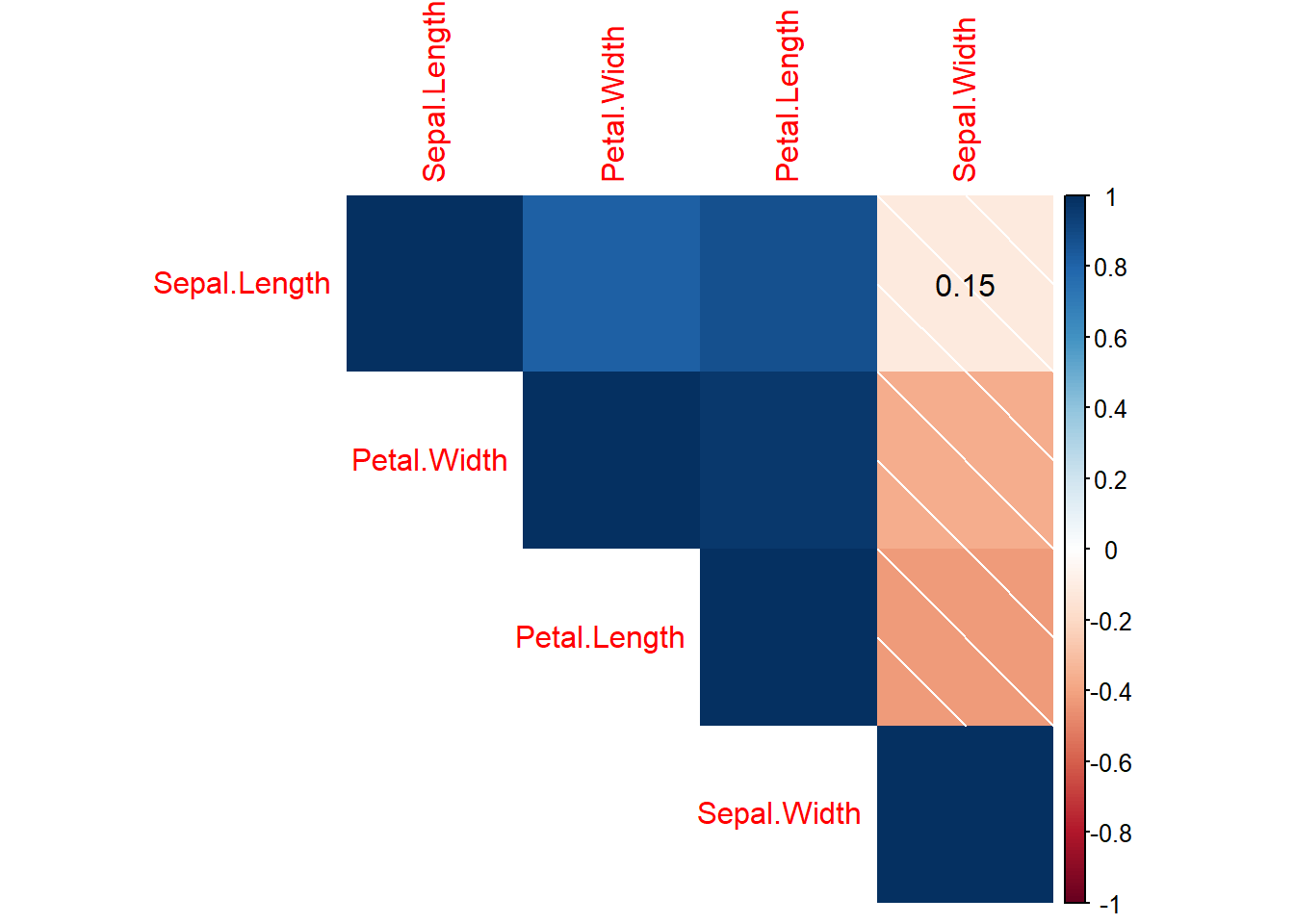
ข้อควรระวัง \(p\)-value ของ Correlation test นั้นอยู่ภายใต้สมมติฐาน (\(t\)-test/\(F\)-test)
\(H_{0}\): ไม่มีความสัมพันธ์เชิงเส้นของทั้งสองตัวแปร (\(r_{xy} = 0\))
\(H_{0}\): มีความสัมพันธ์เชิงเส้นของทั้งสองตัวแปร (\(r_{xy} \neq 0\))
ซึ่งถ้า \(p\) < 0.05 หมายความว่า ท่านมีความมั่นใจมากเพียงพอว่า ความสัมพันธ์เชิงเส้นของทั้งสองตัวแปรนั้น มีมากกว่าการวาดเส้นจากการสร้างจุดแบบสุ่ม ท่านยังคงต้องแปรผลร่วมกับค่า Correlation coefficient ต่อไป
เช่น
\(r_{x,y}\) = 0.2, \(p\) < 0.05 มั่นใจว่ามีความสัมพันธ์เชิงเส้นของสองตัวแปร ความสัมพันธ์เป็นไปในทิศทางเดียวกัน แต่ความสัมพันธ์เชิงเส้นอยู่ในระดับน้อย
\(r_{x,y}\) = 1, \(p\) = 1 ความสัมพันธ์เชิงเส้นอยู่ในระดับดีเยี่ยม แต่ไม่มั่นใจว่ามีความสัมพันธ์กันจริงหรือไม่ เนื่องจากข้อมูลน้อย (เช่น มีข้อมูลเพียงสองจุด \(r_{x,y}\) ย่อมเท่ากับ 1)
\(r_{x,y}\) = 0.3, \(p\) = 1 ความสัมพันธ์เชิงเส้นอยู่ในระดับน้อย แต่ไม่มั่นใจว่ามีความสัมพันธ์กันจริงหรือไม่ เนื่องจากข้อมูลน้อยเกินไป ควรเก็บข้อมูลเพิ่ม
\(r_{x,y}\) = -1, \(p\) < 0.05 มั่นใจว่ามีความสัมพันธ์เชิงเส้นของสองตัวแปร ความสัมพันธ์เป็นไปในทิศทางตรงข้าม และความสัมพันธ์เชิงเส้นอยู่ในระดับดีเยี่ยม
นั่นหมายความว่า ยิ่งจำนวนตัวอย่างเพิ่มขึ้น ความมั่นใจยิ่งเพิ่มขึ้น ไม่ใช่ความสัมพันธ์เพิ่มขึ้น